背景
笔者最近在和医院合作开发一款3D医学影像软件,用的PyQt,其中需要把3D医学影像文件进行可视化,但是在可视化上出现了问题。 这里记录下解决问题的过程。
问题描述
在可视化的过程中,出现了下图所示的问题。
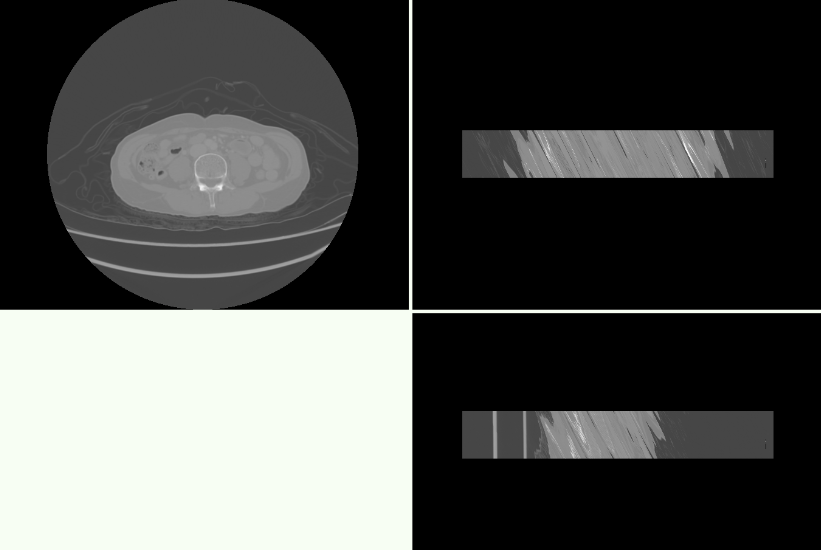
可以看到除了第一个视角正常外,其他视角出现了问题,看样子似乎是图片被压扁了。 然而这并不是数据的问题,如果同样的文件用别的现有的可视化软件打开,是不会出现这些问题的。
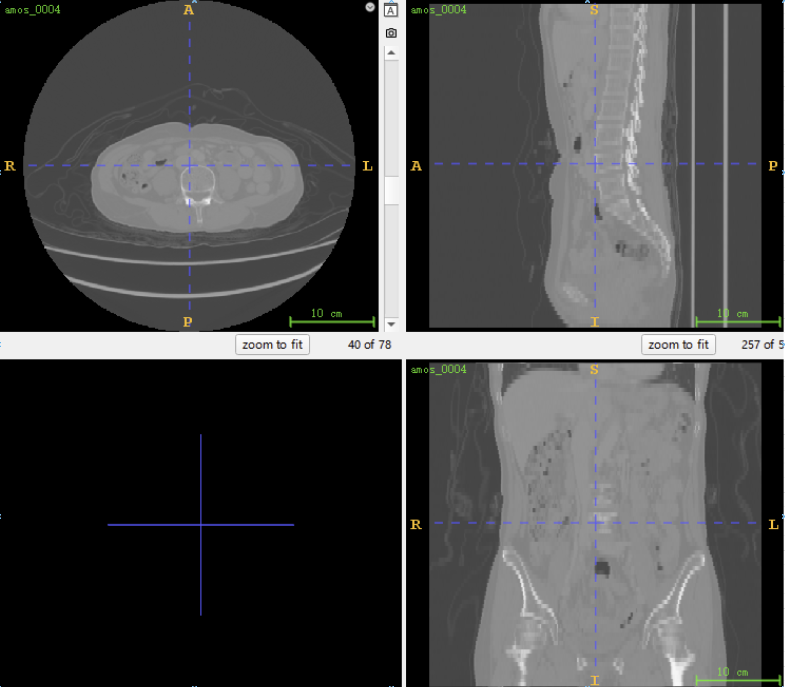
怎么解决
如果我们输出图片的维度会发现,正常视角的图片是(512,512)的,然而其他视角都是(512,149)的。 所以其他视角看上去扁扁的。
data.shape
# (512,512,149)
后来笔者思来想去,决定用ITK-SNAP软件打开图片,放大后发现了问题!
![]()
可以看到正常视角的图片像素是正方形的,而其他视角是长条形的。这就有解决思路了。我们直接把“拉长”图片不就行了 这里使用numpy.repeat,将图片进行“拉长”。于是这个问题就解决了。
data_repeated=[]
for i in range(data.shape[2]):
img = data_arr[:,:,i:i+1].copy()
img = np.repeat(img, 512//149, axis=2)
data_repeated.append(img)
data = numpy.concatenate(data_repeated, axis=2)
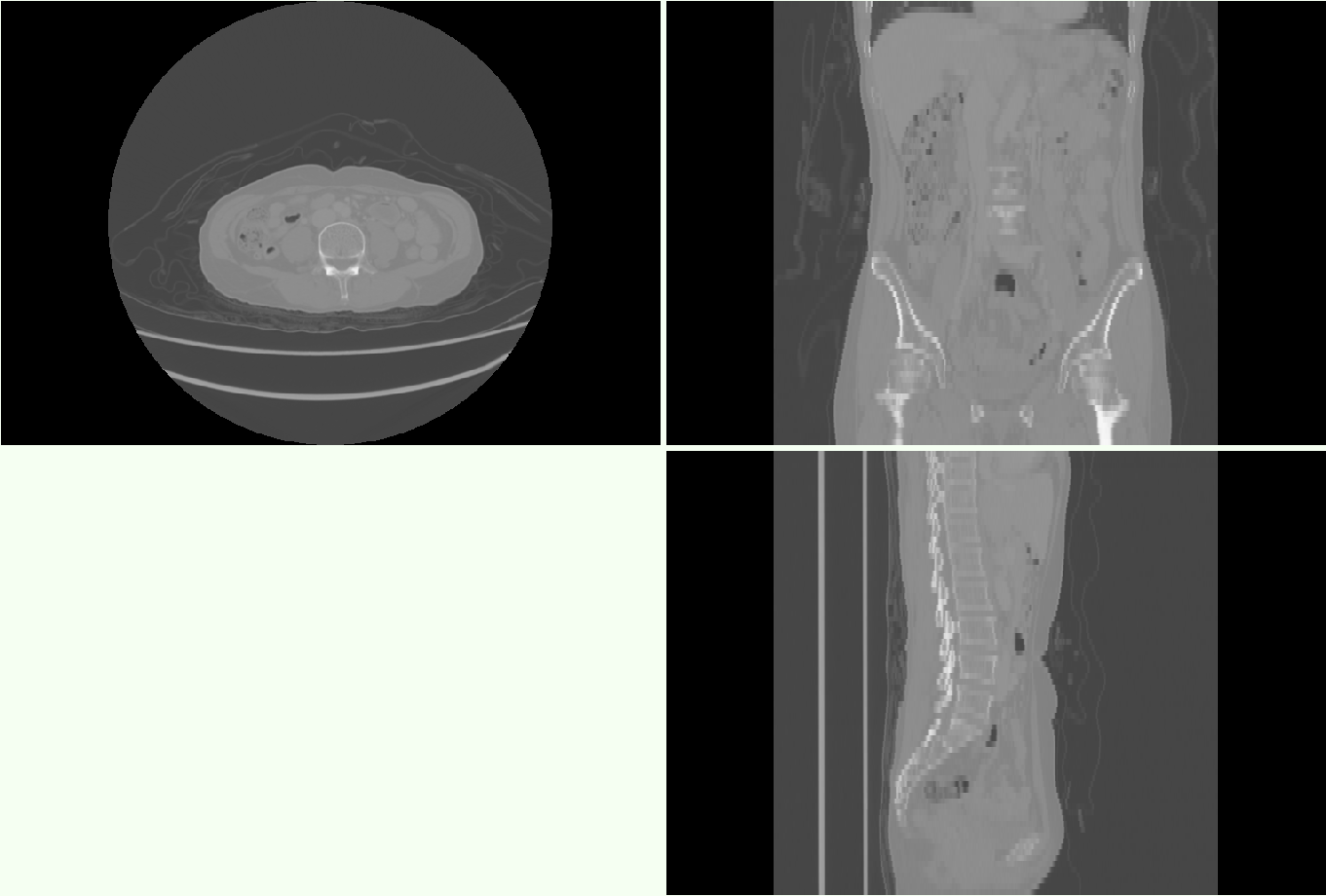
问题的根源
笔者用了简单粗暴的方法直接把图片“拉长”了(512//149)倍,但是我们需要这个问题的通解
我们可以思考,难道ITK-snap也是用这种简单粗暴的方法解决问题的? 我们不妨从gz.nii文件的格式进行入手,这里具体两个网站参考网站
simpleitk官方文档
- https://simpleitk.readthedocs.io/en/master/fundamentalConcepts.html
NIFTI文件格式标准:其记录了nii.gz文件的各种具体信息
- https://brainder.org/2012/09/23/the-nifti-file-format/
在simpleitk官方文档中,我们直接看到了这个问题的全貌。
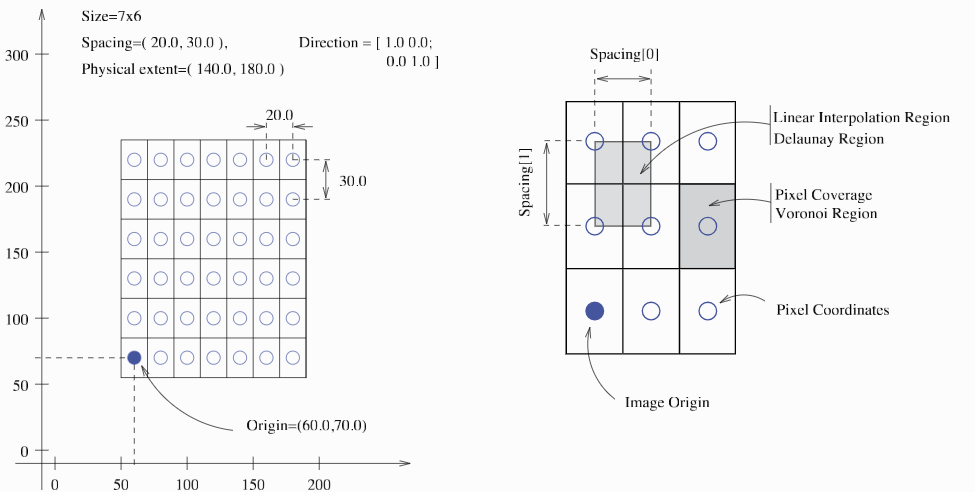 问题的关键就在于图中的Spacing参数,这个参数告诉了我们像素与像素之间的距离,这也是为什么在其他视图中我们会看到长条像素的原因。
问题的关键就在于图中的Spacing参数,这个参数告诉了我们像素与像素之间的距离,这也是为什么在其他视图中我们会看到长条像素的原因。
这个参数在NIFTI文件格式中有具体介绍。其中Spacing参数对应的是NIFTI文件格式中的pixdim,除此之外,xyzt_units参数表示的是单位。 关于这个参数,笔者认为pixdim采样之间的距离,xyzt_units是单位。
我们不妨可以把数据的这些参数输出出来看看:
import nibabel as nib
img = nib.load(data_path)
print(img.header)
# ....
# pixdim : [-1. 0.782 0.782 5. 0. 0. 0. 0. ]
# xyzt_units : 10
# ....
这样我们就知道我们应该具体该怎么“拉长”图像了:
img = np.repeat(img, pixdim[3]//pixdim[1], axis=2)